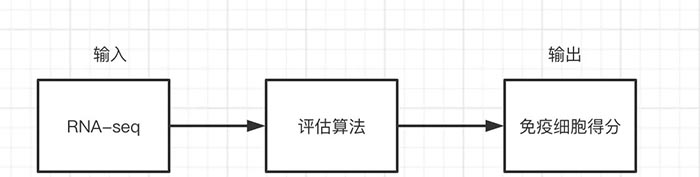
对于RNA-seq的数据,之前我们的分析方法只是局限于单个基因之间的整合分析,最多也就是做一下富集这样的聚类分析。前段时间随着肿瘤免疫的热度,也有人试着开始利用RNA-seq这样的数据来评价患者的免疫情况。
基本的过程也就是我们提供RNA-seq的数据。然后使用相对应的算法来评估每个样本的免疫浸润程度。
目前关于免疫浸润的评估就有 TIMER, CIBERSORT, quanTIseq, xCell, MCP-counter等一些的算法,这些很多都是需要自己下载代码来进行分析的。但是TIMER算法的作者,最近把TCGA所有癌症的RNA-seq的基于不同算法的结果都进行了运算,同时也把结果放到了网站上,这样我们就可以查各个算法当中具体的免疫情况。今天我们就来介绍一个TIMER 2.0 (http://timer.cistrome.org/ ) 这个数据库。
PS:这个数据库有时候运行有点儿慢。如果出不来结果的话,建议科学上网。

这个数据库主要可以进行三个方面的分析:(i) 免疫的相关性分析;(ii)肿瘤基因的扩展(类似于GEPIA的分析);(iii) 上传自己的数据进行免疫情况评估。
免疫相关评价
在免疫相关评价这里,我们可以分析在TCGA当中:1. 基因表达和某一类免疫细胞的相关性; 2. 基因的突变对于和免疫情况的关系; 3. 基因拷贝数的变异和免疫细胞的相关性; 4. 免疫细胞和预后的影响。
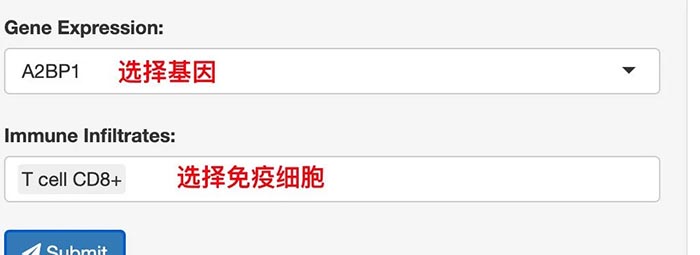
主要操作界面的话,前三种都是选择基因、选择免疫细胞即可。
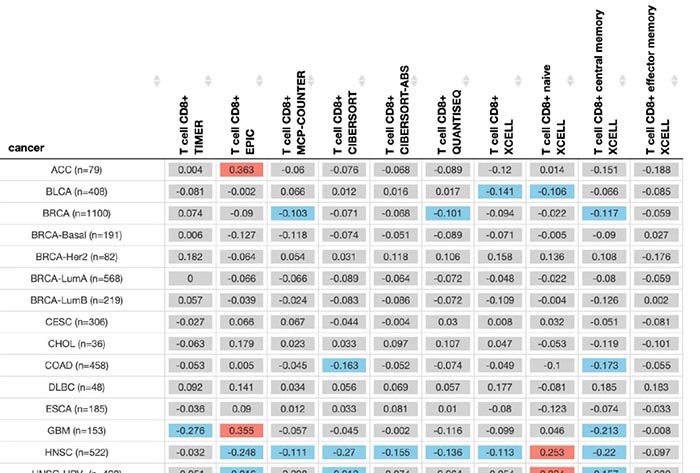
在结果的呈现方面吗,首先数据库会给一个热图来反应不同的免疫浸润评价情况在不同的癌症当的相关性。
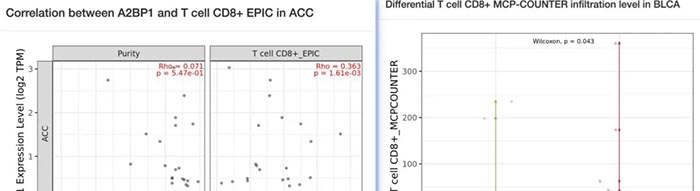
如果我对于某一个结果感兴趣,可以点击其中某一个结果,那么就会绘制具体的结果图形。对于基因表达就是散点图了,对于突变和拷贝数则是小提琴图了。
以上前三种评价的信息。后面的预后主要是来看免疫情况和肿瘤以后的关系了。这个方面,数据库用的是COX回归来进行分析的。由于是COX回归,所以就可以添加其他的矫正因素,这里可以添加临床参数或者某一个基因的表达。
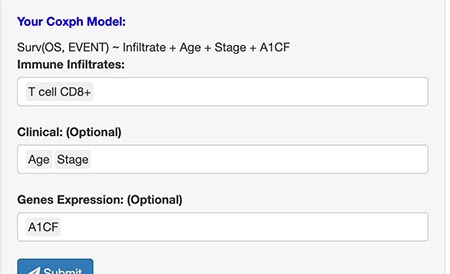
结果的呈现也是首先有一个热图。进一步的我们点击自己感兴趣的就可以看到具体的结果。
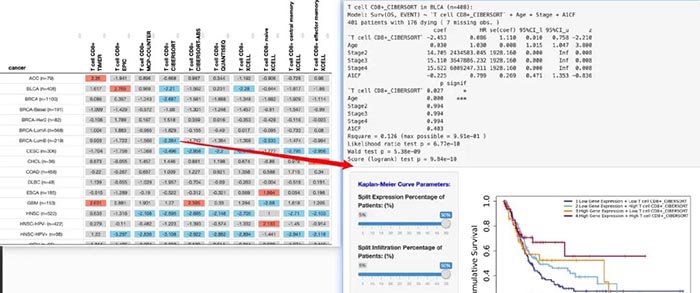
基因相关分析
在这个部分,我们可以查看基因在泛癌当中的结果。主要可以查看:
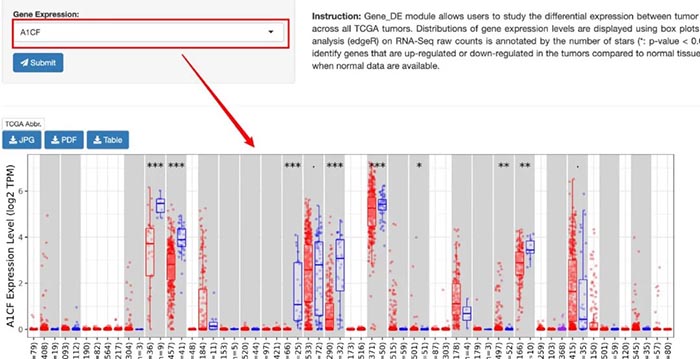
1.基因的差异表达情况。作者使用的是edgr来分析的差异表达结果。我们需要做的就是选择目标基因即可,结果是以箱式图的方式进行呈现的。
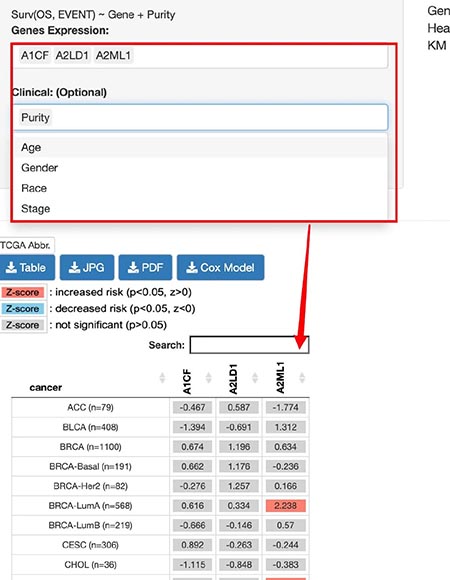
2.基因在泛癌当中的预后情况。在这里我们可以选择多个基因,一次性的查看这些基因的预后情况,同时也添加一些临床参数。
3.突变对于基因的影响。这一部分分析的是是以突变为分组来进行差异表达分析,来分析突变和不突变的组当中基因表达是否有差异。结果是以两组之间的差异表达的log2 fold change来进行热图绘制的。
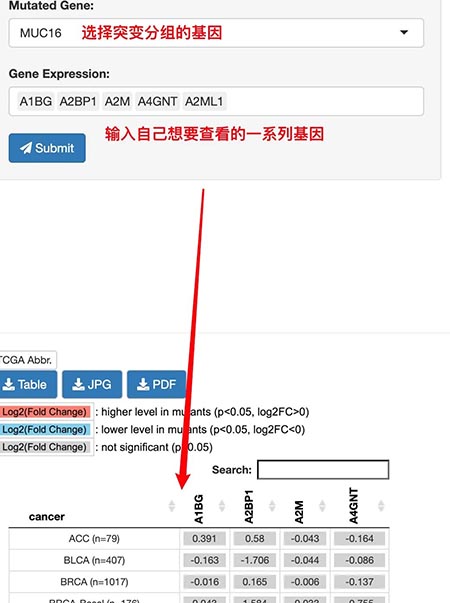
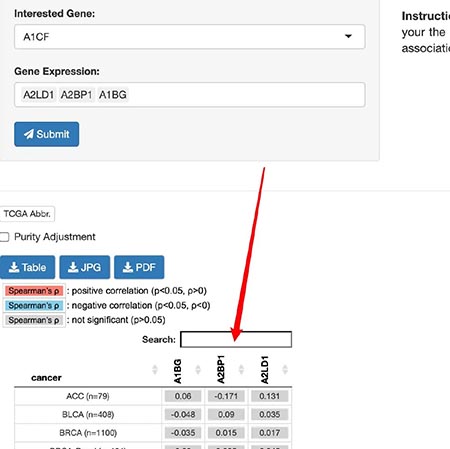
4.查看基因之间的相关性。这里我们可以查看感兴趣的基因和一系列基因的相关性,结果也是通过热图来进行绘制的,每一个位置代表不同癌种的相关性的R2。
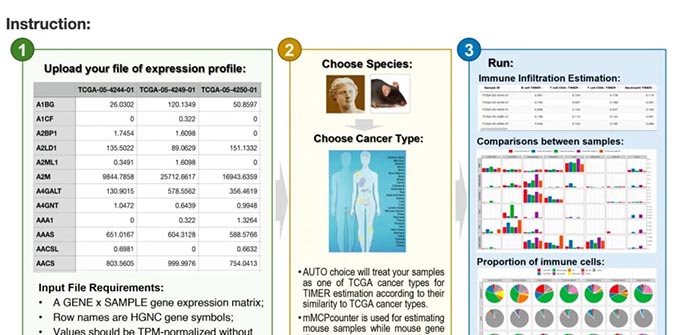
自己数据集免疫情况评估
这个数据库除了可以分析TCGA的现有数据之外,也是可以对自己的数据集进行免疫浸润分析的。刚才我们也提到过,一些关于免疫浸润的算法都是需要代码运行的,因此会需要一定的门槛,所以这个数据库提供了目前的所有算法的评估结果,可以随意下载自己想要算法的结果。
值得注意的是,上传的数据是TPM归一化的数据库。
写到最后
以上就是这个数据库的所有内容了。基本上如果想要做TCGA研究的免疫浸润的话,可以通过这个数据库来查看。主要是汇总了多个算法,所以可以一次的比较,不用再自己进行计算了。














